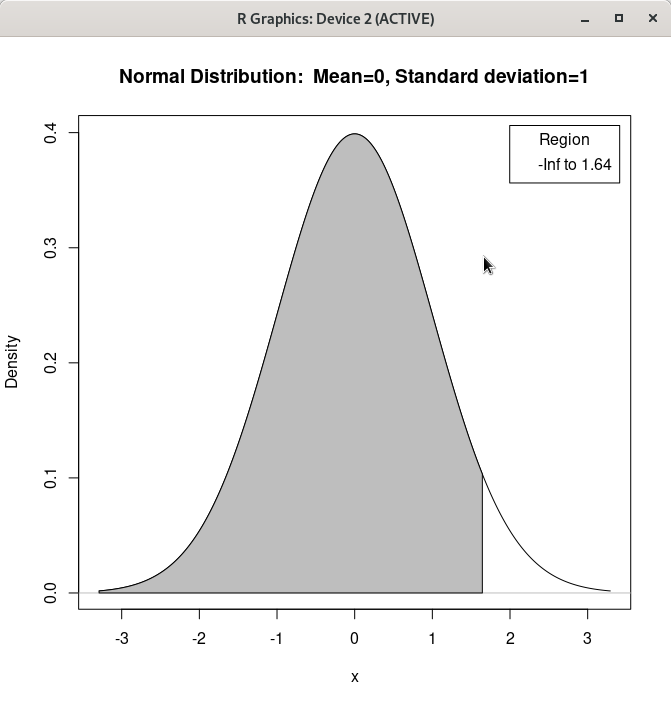
carData::Adler

데이터 > 패키지에 있는 데이터 > 첨부된 패키지에서 데이터셋 읽기... 기능을 선택하면, 위와 같은 메뉴 창을 보게된다.
carData를 선택하여 두번 클릭하면, 오른쪽에 carData 패키지에 내장된 데이터셋 목록이 등장한다. Adler 데이터셋을 선택한다.

data(Adler, package="carData") # Adler 데이터셋 활성화시키기
help("Adler", package="carData")# 도움말파일 열기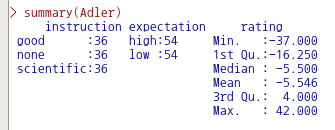
| Adler {carData} | R Documentation |
Experimenter Expectations
Description
The Adler data frame has 108 rows and 3 columns.
The “experimenters” were the actual subjects of the study. They collected ratings of the apparent success of people in pictures who were pre-selected for their average appearance of success. The experimenters were told prior to collecting data that particular subjects were either high or low in their tendency to rate appearance of success, and were instructed to get good data, scientific data, or were given no such instruction. Each experimenter collected ratings from 18 randomly assigned subjects. This version of the Adler data is taken from Erickson and Nosanchuk (1977). The data described in the original source, Adler (1973), have a more complex structure.
Usage
Adler
Format
This data frame contains the following columns:
instruction
a factor with levels: good, good data; none, no stress; scientific, scientific data.
expectation
a factor with levels: high, expect high ratings; low, expect low ratings.
rating
The average rating obtained.
Source
Erickson, B. H., and Nosanchuk, T. A. (1977) Understanding Data. McGraw-Hill Ryerson.
References
Adler, N. E. (1973) Impact of prior sets given experimenters and subjects on the experimenter expectancy effect. Sociometry 36, 113–126.